microbiologie de l’eau
Fin 2012, le manuel suisse des denrées alimentaires (MSDA) a introduit une nouvelle méthode pour évaluer l’état microbiologique de l’eau : le procédé n°333 « Détermination par cytométrie en flux du nombre total de cellules et du rapport entre cellules à forte et faible teneur en acides nucléiques dans l’eau douce ».
analyse TCC & LNA/HNA
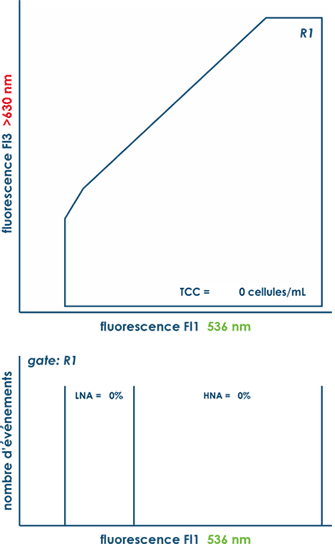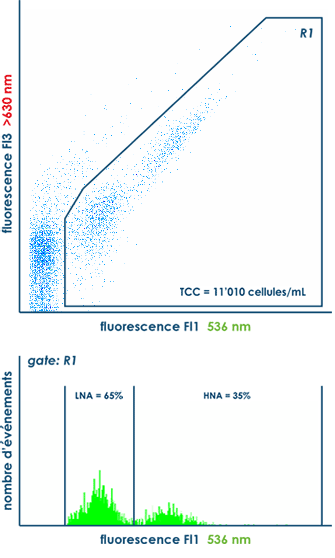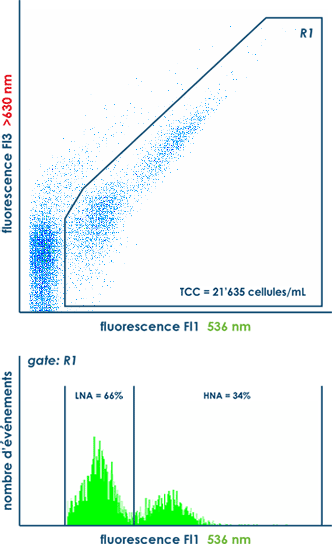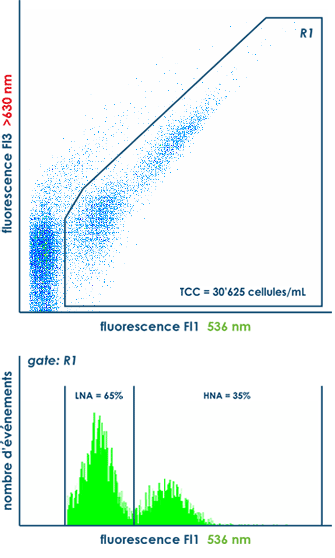 La cytométrie en flux (FCM, flow cytometry) permet de quantifier – en 20 minutes seulement – le nombre total de cellules (TCC, total cell count) présentes dans un échantillon d’eau. En parallèle, la méthode permet aussi de déterminer les fractions de cellules à faible (LNA, low nucleic acid) et forte (HNA, high nucleic acid) teneur en acides nucléiques.
La cytométrie en flux (FCM, flow cytometry) permet de quantifier – en 20 minutes seulement – le nombre total de cellules (TCC, total cell count) présentes dans un échantillon d’eau. En parallèle, la méthode permet aussi de déterminer les fractions de cellules à faible (LNA, low nucleic acid) et forte (HNA, high nucleic acid) teneur en acides nucléiques.
Le nombre total de cellules et les fractions de cellules à faible et forte teneur en acides nucléiques constituent une véritable « empreinte digitale » de la communauté microbiologique et sont typiques à chaque eau. Contrairement aux méthodes traditionnelles, l’analyse TCC & LNA/HNA donne donc une description réaliste et complète de l’état microbiologique de l’eau.
Une variation – spatiale et/ou temporelle – de l’un de ces paramètres est synonyme d’une instabilité de l’état microbiologique de l’eau. Elle peut être signe d’une contamination ou d’un problème technique dans l’installation de traitement ou dans le réseau de distribution d’eau potable.
analyse ICC/DCC (Live/Dead)
 En complément à l’analyse TCC & LNA/HNA, la cytométrie en flux permet également de fournir des renseignements sur les fractions de cellules intactes (ICC, intact cell count, ou Live) et endommagées (DCC, damaged cell count, ou Dead). Cette analyse est particulièrement intéressante pour évaluer l’efficacité des procédés de potabilisation de l’eau, simples ou complexes.
En complément à l’analyse TCC & LNA/HNA, la cytométrie en flux permet également de fournir des renseignements sur les fractions de cellules intactes (ICC, intact cell count, ou Live) et endommagées (DCC, damaged cell count, ou Dead). Cette analyse est particulièrement intéressante pour évaluer l’efficacité des procédés de potabilisation de l’eau, simples ou complexes.
applications
- évaluation spatiale et temporelle de la stabilité / activité microbiologique de l’eau potable dans le réseau de distribution
- évaluation de l’état microbiologique de l’eau lors des différents procédés de potabilisation
- détection de phénomènes de (re)croissance et de contamination microbiologique
- évaluation de l’impact des eaux de surface sur la qualité microbiologique des eaux souterraines
principe du procédé n°333 par FCM
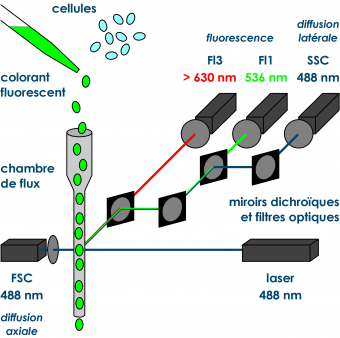
Les cellules présentes dans un échantillon d’eau sont marquées avec un colorant fluorescent se liant à l’ADN. L’échantillon est ensuite introduit dans le cytomètre en flux, où les cellules et d’autres particules subissent une focalisation hydrodynamique par un liquide vecteur dans la chambre de flux. Celles-ci, passant une à une le faisceau lumineux du laser, diffusent la lumière et émettent de la fluorescence. Ces signaux sont ensuite séparés par un jeu de miroirs dichroïques et filtres optiques, collectés par des photomultiplicateurs ou photodiodes, puis amplifiés et numérisés.
La cytométrie en flux est un procédé multiparamétrique. Elle attribue à chaque cellule / particule les différents signaux optiques émis, c’est-à-dire la diffusion axiale (FSC, forward scatter) et latérale (SSC, sideward scatter) et la fluorescence à différentes longueurs d’ondes (notamment Fl1 à 536±40 nm et Fl3 à >630 nm). Elle peut déceler, analyser et traiter plusieurs centaines à quelques milliers d’événements par seconde. Par une combinaison des paramètres enregistrés, il est possible de distinguer les cellules des particules inorganiques et donc ainsi d’énumérer rapidement le nombre total de cellules et de discriminer les fractions LNA/HNA et Live/Dead.
cytométrie en flux vs. inoculation en gélose
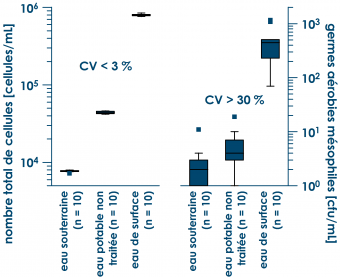
Une grande majorité des microorganismes dans l’eau est viable mais non-cultivable. Par conséquent, il est généralement admis que moins de 0.1 % des cellules bactériennes présentes dans un échantillon d’eau sont décelées lors des analyses classiques par ensemencement en milieu gélosé (p. ex. l’analyse des germes aérobies mésophiles). Contrairement à la cytométrie en flux, ces techniques traditionnelles donnent donc une image très incomplète de l’état microbiologique de l’eau.
Outre la rapidité de l’analyse par cytométrie en flux (env. 20 minutes contre minimum 72 h pour les analyses traditionnelles), cette technique est également très fiable. En effet, l’évaluation de la reproductibilité et de la répétabilité des mesures a montré des coefficients de variation inférieurs à 3 %.